Un article du doctorant en biochimie et récipiendaire d’une bourse Vanier Philippe Després, publié dans le journal Nature Communications, démontre que cela est maintenant possible grâce à la biologie synthétique.
La biologie synthétique a pour but de modifier les génomes pour en étudier la fonction et en construire des nouvelles en s’inspirant de ce que l’évolution a construit.
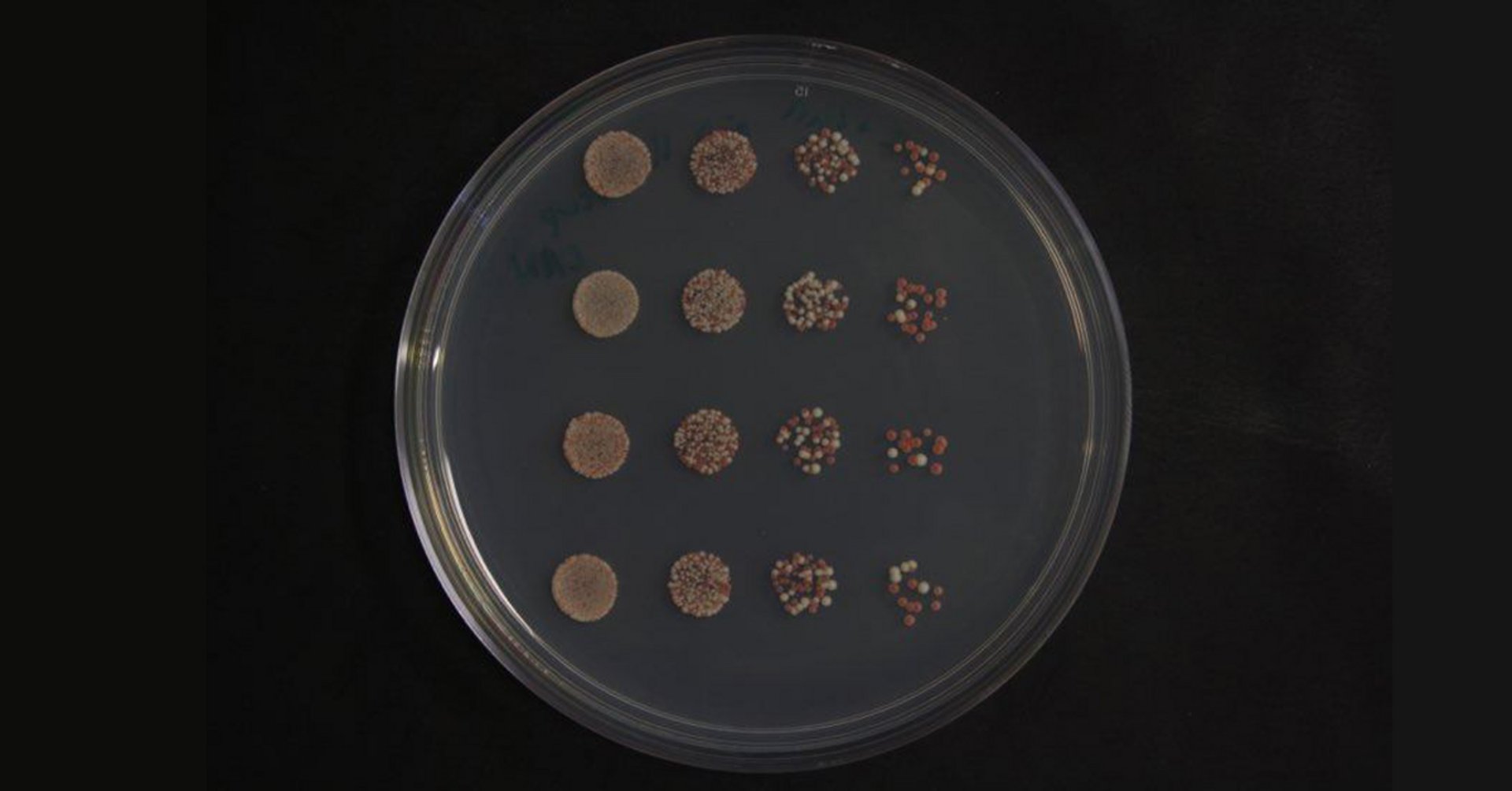
Les éditeurs de base dérivés des systèmes CRISPR-Cas9 et des enzymes d'édition d’ADN sont des outils en or pour l’édition de gènes avec précision : ils fonctionnent en effaçant des parties d’un gène et en réécrivant leur contenu. Ils permettent donc non seulement de voir quelle est l’importance des gènes, mais aussi celle de parties de gènes spécifiquement. Cette approche a le potentiel d'augmenter la résolution de la génétique par plusieurs milliers de fois. Toutefois, jusqu’à récemment, ces outils d’édition n’avaient pas été testés à l’échelle génomique.
Dans cette étude intitulée «Perturbing proteomes at single residue resolution using base editing», l’équipe de Christian Landry, professeur à la Faculté et chercheur à l’Institut de biologie intégrative et des systèmes (IBIS), a testé la capacité de l'outil d’édition génétique de base Target-AID à modifier systématiquement les gènes à l'échelle du génome. Ils ont procédé en ciblant les gènes essentiels de levure à vin.
Environ 17 000 sites individuels en parallèle ont été mutés sur plus de 1 500 gènes, et plus de 700 sites où les mutations ont un impact significatif sur la croissance ont été identifiés, leur permettant ainsi d’affirmer que l'édition de base est un outil puissant pour identifier les résidus clés de centaines de protéines simultanément.
Soulignons également la collaboration d'Alexandre K. Dubé, diplômé en biochimie et professionnel de recherche dans l’équipe du Dr Landry. Ces travaux impliquent aussi des collaborateurs de l’Université de Tokyo.
Nous vous invitons à lire l’article à ce sujet.
14 mai 2020